不均衡データのダウンサンプリングについて
論文や学術上では、不均衡データのときはダウンサンプリングしてデータを揃えたほうがいい、と言われるのが一般的かと思う。が、実際に分析をしてきた経験上、ダウンサンプリングすると特徴がうまく出ないことが多い。サンプリングせず、不均衡のままでモデルを作った方が正解データの特徴がはっきり出る。
そもそもダウンサンプリングすると予測力がどうなるのか?と気になって、Rで簡単なシミュレーションをしてみたところ、確かにダウンサンプリングしても予測力は変わらないし、特徴はちゃんと出た(詳細は下記)。ただ、実際のデータは変数が数千〜数万あったり、スパースだったりするので、サンプリングすることで特徴が出にくくなるのかもしれないと推察。スパースデータでのダウンサンプリングとかのテーマで研究もあるのかもしれない。あと、正解データの特徴が何パターンもあってサンプルサイズも多くないときは、非正解データをサンプリングすると特徴が出にくくなることもあるかも?(これもスパースなのが主な原因かな
サンプリングしない状態が真の状態であることは念頭に置きつつ、単純な特徴だとサンプリングしても大丈夫、サンプリングでうまく特徴が出なかったらサンプルサイズを増やしたりなるべくサンプリングせずに分析してみる、ということかな、といまのところは理解しておく。多重共線性も学術上はうるさく言われるけど実務上は気にすることはないし(共線性の影響が大きい重回帰はもうほとんど使わない)、学術と実務の違いなのかも。
※シミュレーション内容
・正解データ1,000
・非正解データ100,000
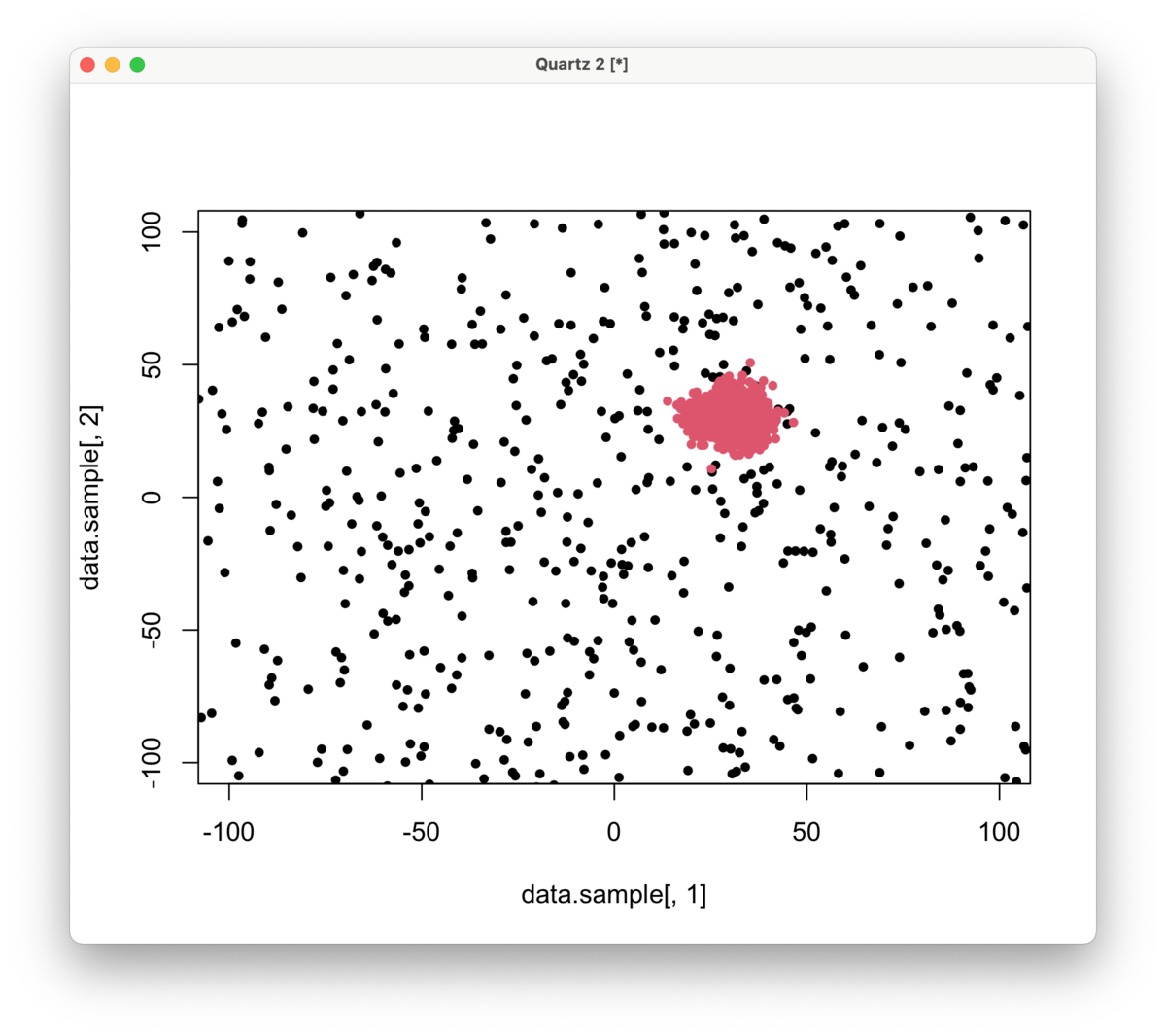
赤:正解データ
黒:非正解データ(1,000サンプリング)
サンプリングしない結果


AUC:0.9842543
1:1ダウンサンプリングした結果

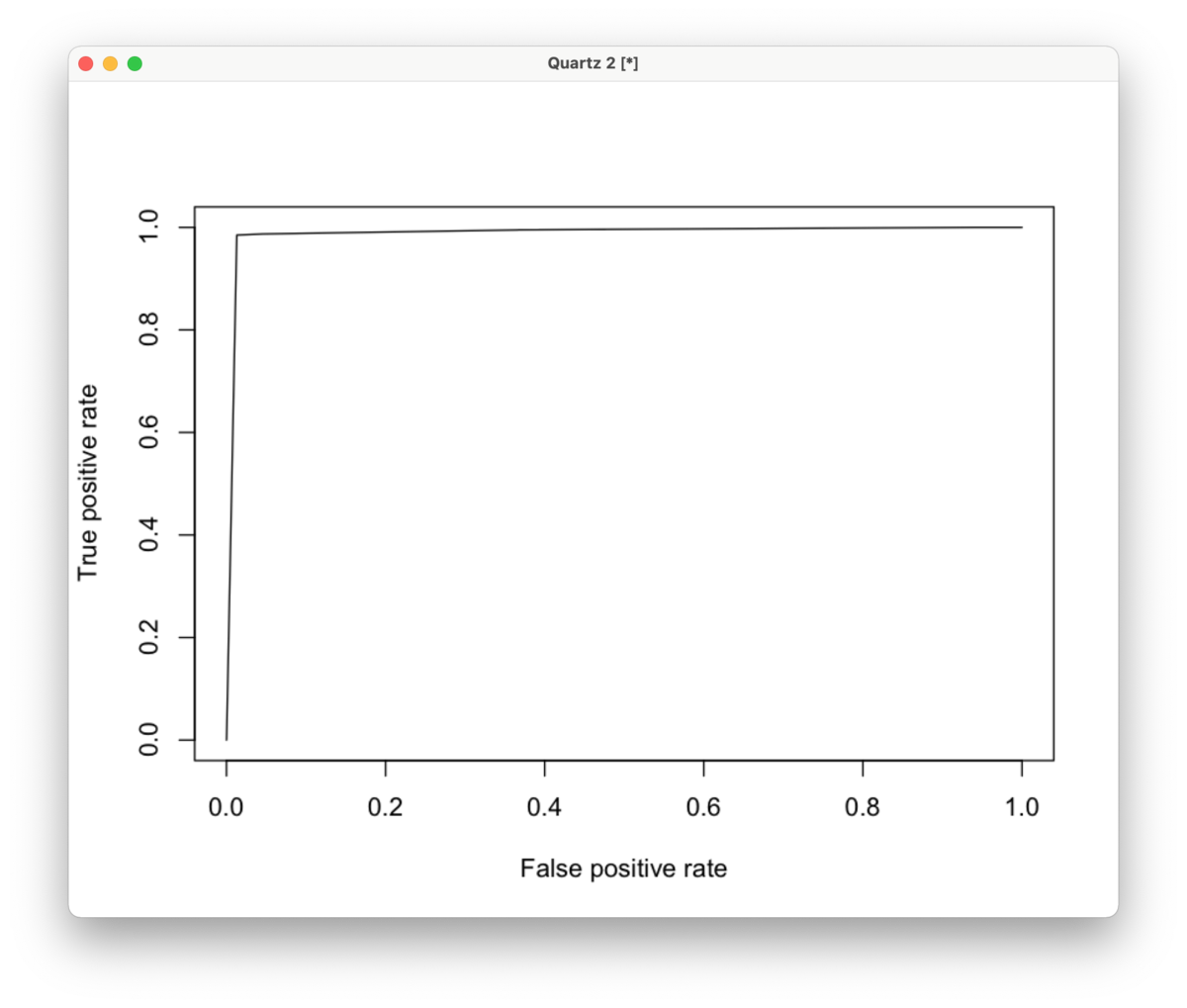
AUC:0.9886305
※下記Rコード
data1 <- cbind(x=rnorm(100000)*100, y=rnorm(100000)*100, flag=0)
data2 <- cbind(x=rnorm(1000)*5+30, y=rnorm(1000)*5+30, flag=1)
data <- rbind(data1, data2)
#plot(data[, 1], data[, 2], col=data[, 3]+1, pch=20, xlim=c(-100, 100), ylim=c(-100, 100))
library(rpart)
library(rpart.plot)
library(partykit)
library(ROCR)
tree <- rpart(factor(data[, 3]) ~ data[, 1] + data[, 2])
plot(tree)
text(tree, use.n = TRUE)
rpart.plot(tree)
plot(as.party(tree), gp=gpar(fontsize=9))
pred <- predict(tree)[(nrow(data)+1):(nrow(data)*2)]
pred1 <- prediction(pred, data[, 3])
perf <- performance(pred1,"tpr","fpr")
plot(perf)
auc.tmp <- performance(pred1,"auc")
auc <- as.numeric(auc.tmp@y.values)
auc
data.sample <- rbind(data1[sample(1:100000, 1000), ], data2)
#plot(data.sample[, 1], data.sample[, 2], col=data.sample[, 3]+1, pch=20, xlim=c(-100, 100), ylim=c(-100, 100))
tree.sample <- rpart(factor(data.sample[, 3]) ~ data.sample[, 1] + data.sample[, 2])
plot(as.party(tree.sample), gp=gpar(fontsize=9))
pred <- predict(tree.sample)[(nrow(data.sample)+1):(nrow(data.sample)*2)]
pred1 <- prediction(pred, data.sample[, 3])
perf <- performance(pred1,"tpr","fpr")
plot(perf)
auc.tmp <- performance(pred1,"auc")
auc <- as.numeric(auc.tmp@y.values)
auc