コロナ陽性者数に正規関数を当てはめた推測の評価2
推定を作ってから約2ヶ月経ってからのコロナ陽性者数の推測結果、以下になりました。

第5波までの傾向だったらもう収束しているはずですが、収束しておらず、1日6,000〜7,000人くらいで停滞している感じです。これくらいの陽性者数が底になっている印象。恐らく、これからは今までと違う動きをすると思われるので、また違う推測モデルが必要かも。
コロナ陽性者数に正規関数を当てはめた推測の評価
2/6に、↓の記事で東京都のコロナ陽性者数に正規関数を当てはめて推移を予測しました。1ヶ月ほど経ったので、その予測の評価をしてみました。
評価した結果は次の図のようになりました。

オレンジ線が実際の陽性者数で、青破線が2/6までのデータを使ったモデルで推測した値です。モデルを作ってから少し陽性者数が増加し、推測ではもっと増えると計算されてましたが、そこまで増えませんでした。ただ、その後の減り方は緩やかだったので、3/14時点では正規曲線の値に近づいています。
この後どうなっていくか分かりませんが、モデルだと4/10あたりで収束になっています。ただ実際のオレンジ線はゆっくり減ってくように見えるので、収束はもっと遅い可能性もあります。
2月の中旬くらい、モデルほど実数が増えなかった原因としては、個人的な感想ですが、感染者数に検査が追いついてなかったのではと思ったり、みなし陽性で実際の陽性者より少なく報告されてるなどあるのかなと思ったりしています。何かエビデンスがあるわけでないので、純粋な個人的感想です。
1/4あたりに第6波が始まり、2/6までの1ヶ月くらいのデータで計算したモデルですが、全く外れてるというわけではない感じがします。流行の周期自体もだいたい3ヶ月くらい。なぜかは分かりませんが、集団行動とか免疫とかが関係しているのかな、?4月上旬あたり、収束しているといいです。
計算上の最適とビジネス上の最適の違いについて
AI、機械学習、データサイエンスでモデルを作って分析したり予測するとき、モデルをどう作ればいいか?という問題で。
学問的には、交差検証(クロスバリデーション)をしたり、何らかの当てはまり指標を最適化してモデルを作ったりすることが望ましいとされます。
が、自分がこれまでデータをビジネス実績に結びつけてきた実感としては、そうやってつくったモデルは役に立たないことが多い。
全体的に、保守的なモデルになり、業務知見と同じような結果で、モデル使う必要ない、データでは何も見つからないね、となってしまうことが多い。業務知見を超える知見は、最適なモデルより"少し過学習気味"に設定したときに現れてるように思っています。
自分が新規のデータを分析するときは、だいたい決定木を試してみるが、わざと過学習気味の木を作る。そしてそれをビジネス側に説明しながら、実際の業務でどう活用できるかを詰めていく。
決定木の場合で言うと、枝の2〜3段目くらいまでは業務知見で説明が付く事が多い。さらに分岐させていくと、業務知見ではカバーできてなかった細かいルールになっていく。そのルールが、ほんとうに法則を表しているのか、それともたまたまの過学習なのか。それは最適化の計算結果で判断するのではなく、ビジネス側と議論してお互い一緒に考える。
最終的には、実際に使ってみないとそのルールが正しいかどうかは判断できないので、間違っているかもしれないということを念頭におきながら、試せる範囲内で実証実験をしていく。それが、最速で最大限に分析結果をビジネス成果に繋げることができる方法かなと。
データを活用して実際のビジネス収益を上げられるかどうかは、計算上の最適解よりももう少し深いところの特徴が有効なものかどうか次第かなと感じます。
コロナ陽性者数の推移への正規関数の当てはめ
コロナの感染者数の推移に関して、感染拡大が上がって下がる変化の仕方を関数で表現できないか、またそれによってある程度拡大は収束の推測ができないかと思っていました。自分が知っている関数の中では、正規分布の確率関数が、拡大と収束の形に一番近いため、正規関数に当てはめて分析してみました。
※この取り組みは、私個人の見解で行っているものであり、また厳密な研究をしているわけではないので、必ずしも正解というわけではなく、あくまでも参考例として捉えてもらえればと思います。
結果的にいうと、正規関数のあてはまりは悪くなく、一定程度の参考になりそうかなというものです。東京の感染者数に対して、いま感染拡大している第6波に当てはめてみると、第6波が始まってから52日程度(2/24あたり)がピークで、7日平均が3.5万人程度という計算になっています(本記事一番下↓の図参考)。7日平均が約3.5万人なので、曜日ピークの水・木曜あたりは4〜5万人くらい?→パラメータを追加して分析したら47日後(2/19あたり)に約2.7万人という結果に
ただこの計算も、この1週間で結構ぶれているので、また数日や1週間程度したら大きく変わるかもしれません。第6波とみられる日から21日間のデータを使って計算したピーク人数は約6.6万人、28日間のデータを使ったピーク人数は約3.3万人と大きく減少しました。この1週間くらい、増加スピードが緩やかになっている影響かと思います。
今回行った計算は、”増えだしてからx日間のデータを使って正規関数を当てはめ、もっとも当てはまりがいいパラメータを選択する”、というものです。この方法を東京の第4波と第5波のデータに当てはめてみたところ、そこそこ当てはまりが良かったです。第4波のときは実際のピークが935人(過去7日間の平均)であるのに対し、推定値が961人〜1,022人。第5波のときは4,923人(同上)であるのに対し、4024人〜7,347人(35〜42日の間で拡大スピードが少し下がった)でした。
この方法が、全国のデータで通用するのか、また世界各国のデータで通用するのかは未確認です。下記、Rで行ったプログラムと、結果の一部です。データは、下記の東京都のオープンデータから取得したcsvを利用しています。
データ取得元:東京都 新型コロナウイルス陽性患者発表詳細 - 東京都_新型コロナウイルス陽性患者発表詳細(全期間一括版) - 東京都オープンデータカタログサイト
■第4波を2021/3/18〜6/18としてデータを作成したときの結果。
・3/18から21日(3週間)のデータを使って計算した結果

縦軸↑:7日平均感染者数
横軸→:開始日からの日数(このグラフは3/18が1、50の時点は5/6)
丸:観測された値
青丸:計算に使ったデータ
黒丸:その後実際に観測されたデータ
曲線:計算した正規関数
・3/18から56日(8週間)のデータを使って計算した結果

■第5波を2021/7/5〜10/6としてデータを作成したときの結果。
・21日間のデータを利用

・28日間のデータを利用(この時点ではピークが大きく計算されている)

・35日間のデータを利用(28〜35日間の拡大が少なかったためか、ピークも小さくなった)

・70日間のデータを利用(若干当てはまりが悪く見える。正規関数でなく、歪正規関数のように、他の関数の方が当てはまりがいいのかもしれない)

■第6波を2022/1/4〜としてデータを作成したときの結果。最大で2/5までの33日間のデータを利用。
・21日間のデータを利用

・28日間のデータを利用(感染者が急に増えたためか、ピークが約6.6万人と計算)

・33日間のデータを利用(この1週間くらいは感染拡大が鈍化したためか、ピークが約3.5万人に下がる。2/24日あたりをピークに減少に向かう?拡大の鈍化が続けば、ピークの人数ももっと減るかも)

↓追記:パラメータ追加して計算するとピークまでの日数とピーク人数が減少(2/19あたりがピーク、7日間平均が約2.7万人、水・木のピークだと3.5〜4万人くらい?)。

上に書いたように、本記事は正確な研究に基づいたものではないですし、流行学的な感染モデルのようなものを仮定したりしているわけではありません。ただ観測された感染者数だけを使って計算しています。今後の個々人の行動や政策などによって大いに変わる可能性はありますが、ある程度の参考や目安になればと思っています。
Data <- read.csv(".../130001_tokyo_covid19_patients.csv", as.is=T) table(Data$発症_年月日) Kansensha <- table(Data$公表_年月日) # 7日間移動平均を利用→正規関数のブレが少なくなった Kansensha2 <- filter(Kansensha, rep(1, 7))/7 plot(Kansensha) plot(Kansensh2) Prm <- NULL # 調査するパラメータの設定。当てはまりが良さそうな値の周辺を設定している # 他の地域や時期を分析するときにはパラメータが大きく変わる可能性はある peak <- c(100, 200, 300, 500, 1000, 3000, 5000, 10000, 30000, 50000) mean <- c(45, 49, 50, 51, 52, 53, 55) sigma <- c(0.05, 0.054, 0.055, 0.056, 0.057, 0.058, 0.059, 0.06) for(a in 1:length(peak)){ for(b in 1:length(mean)){ for(c in 1:length(sigma)){ Prm <- rbind(Prm, c(peak[a], mean[b], sigma[c])) } } } head(Prm) dim(Prm) Ryukou4 <- Kansensha[391:483] Ryukou5 <- Kansensha[500:593] Ryukou6 <- Kansensha[683:714] R <- Ryukou3 Len <- length(R) Len <- 40 Est <- function(R, Len){ # base <- mean(R[1:7]) base <- min(R[1:7]) Ryukou <- R - base SDS <- NULL # i <- 1 for(i in 1:nrow(Prm)){ DS <- NULL for(x in 1:Len){ peak <- Prm[i, 1] mean <- Prm[i, 2] sigma <- Prm[i, 3] # sigmaが小さい方が分布の幅が大きくなる結果になったので、式のプラスマイナスや分母が間違ってる可能性? # ただ、yを計算してプロットしたら当てはまりの良い正規分布になっているので、結果には問題なさそう DS <- c(DS, (Ryukou[x] - peak * 1/(2*pi*sigma^2)^0.5 * exp(- (x - mean)^2 / 2*sigma^2))^2) } SDS <- c(SDS, sum(DS)) # print(i) } # SDS print(min(SDS)) print(Prm[(SDS == min(SDS)), ]) print(max(R)) x <- 1:(length(Ryukou)+100) prm <- Prm[(SDS == min(SDS)), ] peak <- as.numeric(prm[1]) mean <- as.numeric(prm[2]) sigma <- as.numeric(prm[3]) # sigma <- 0.1 # peak <- 200 y <- base + peak * 1/(2*pi*sigma^2)^0.5 * exp(- (x - mean)^2 / 2*sigma^2) print(max(y)) plot(R, ylim=c(0, max(y*1.1)), xlim=c(0, max(x))) points(R[1:Len], col="blue") lines(y) y } Ryukou4.2 <- Kansensha2[391:483] Ryukou5.2 <- Kansensha2[500:593] Ryukou6.2 <- Kansensha2[683:715] Est(Ryukou4, 21) Est(Ryukou4, 35) Est(Ryukou4, 42) y4.2.21 <- Est(Ryukou4.2, 21) y4.2.35 <- Est(Ryukou4.2, 35) y4.2.42 <- Est(Ryukou4.2, 42) y4.2.49 <- Est(Ryukou4.2, 49) y4.2.56 <- Est(Ryukou4.2, 56) y4.2.63 <- Est(Ryukou4.2, 63) y4.2.70 <- Est(Ryukou4.2, 70) y5.2.21 <- Est(Ryukou5.2, 21) y5.2.35 <- Est(Ryukou5.2, 35) y5.2.42 <- Est(Ryukou5.2, 42) y5.2.42 <- Est(Ryukou5.2, 70) y6.2.14 <- Est(Ryukou6.2, 14) y6.2.21 <- Est(Ryukou6.2, 21) y6.2.28 <- Est(Ryukou6.2, 28) y6.2.33 <- Est(Ryukou6.2, 33)
パラメータ追加
peak <- c(100, 200, 300, 500, 1000, 3000, 4000, 5000, 6000, 7000, 10000, 30000, 50000) mean <- c(44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 55, 60, 70) sigma <- c(0.01, 0.05, 0.054, 0.055, 0.056, 0.057, 0.058, 0.059, 0.06, 1) for(a in 1:length(peak)){ for(b in 1:length(mean)){ for(c in 1:length(sigma)){ Prm <- rbind(Prm, c(peak[a], mean[b], sigma[c])) } } } head(Prm) dim(Prm)
決定木のプログラム
決定木のプログラムメモ
library(rpart)
library(ROCR)
Data <- read.csv("...", as.is=T)
head(Data)
names(Data)
dim(Data)
tree01 <- rpart(Flag ~ ., data=Data, control=rpart.control(cp = 0.05))
par(mar=c(2, 2, 2, 2))
plot(tree01)
text(tree01, use.n = TRUE)
tree01
sink("...")
tree01
sink()
# 木の性能チェック
pred <- 1 - predict(tree01)[1:nrow(Data)]
table(pred, Data$Flag)
prediction <- prediction(pred, factor(Data$Flag))
roc <- performance(prediction, "tpr", "fpr")
par(mar=c(3, 3, 3, 3))
plot(roc)
auc <- performance(prediction, "auc")
as.numeric(auc@y.values)
不均衡データのダウンサンプリングについて
論文や学術上では、不均衡データのときはダウンサンプリングしてデータを揃えたほうがいい、と言われるのが一般的かと思う。が、実際に分析をしてきた経験上、ダウンサンプリングすると特徴がうまく出ないことが多い。サンプリングせず、不均衡のままでモデルを作った方が正解データの特徴がはっきり出る。
そもそもダウンサンプリングすると予測力がどうなるのか?と気になって、Rで簡単なシミュレーションをしてみたところ、確かにダウンサンプリングしても予測力は変わらないし、特徴はちゃんと出た(詳細は下記)。ただ、実際のデータは変数が数千〜数万あったり、スパースだったりするので、サンプリングすることで特徴が出にくくなるのかもしれないと推察。スパースデータでのダウンサンプリングとかのテーマで研究もあるのかもしれない。あと、正解データの特徴が何パターンもあってサンプルサイズも多くないときは、非正解データをサンプリングすると特徴が出にくくなることもあるかも?(これもスパースなのが主な原因かな
サンプリングしない状態が真の状態であることは念頭に置きつつ、単純な特徴だとサンプリングしても大丈夫、サンプリングでうまく特徴が出なかったらサンプルサイズを増やしたりなるべくサンプリングせずに分析してみる、ということかな、といまのところは理解しておく。多重共線性も学術上はうるさく言われるけど実務上は気にすることはないし(共線性の影響が大きい重回帰はもうほとんど使わない)、学術と実務の違いなのかも。
※シミュレーション内容
・正解データ1,000
・非正解データ100,000
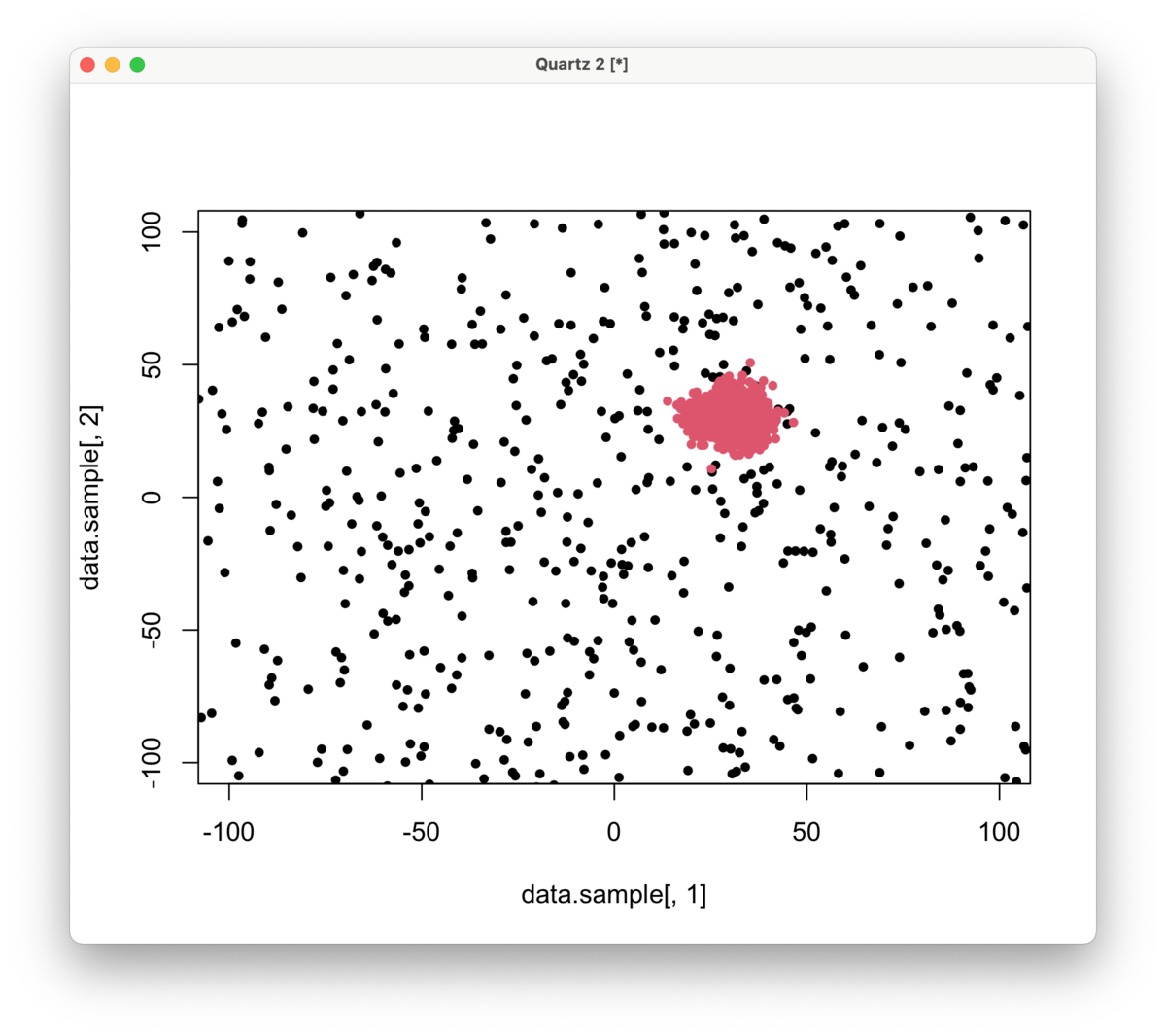
赤:正解データ
黒:非正解データ(1,000サンプリング)
サンプリングしない結果


AUC:0.9842543
1:1ダウンサンプリングした結果

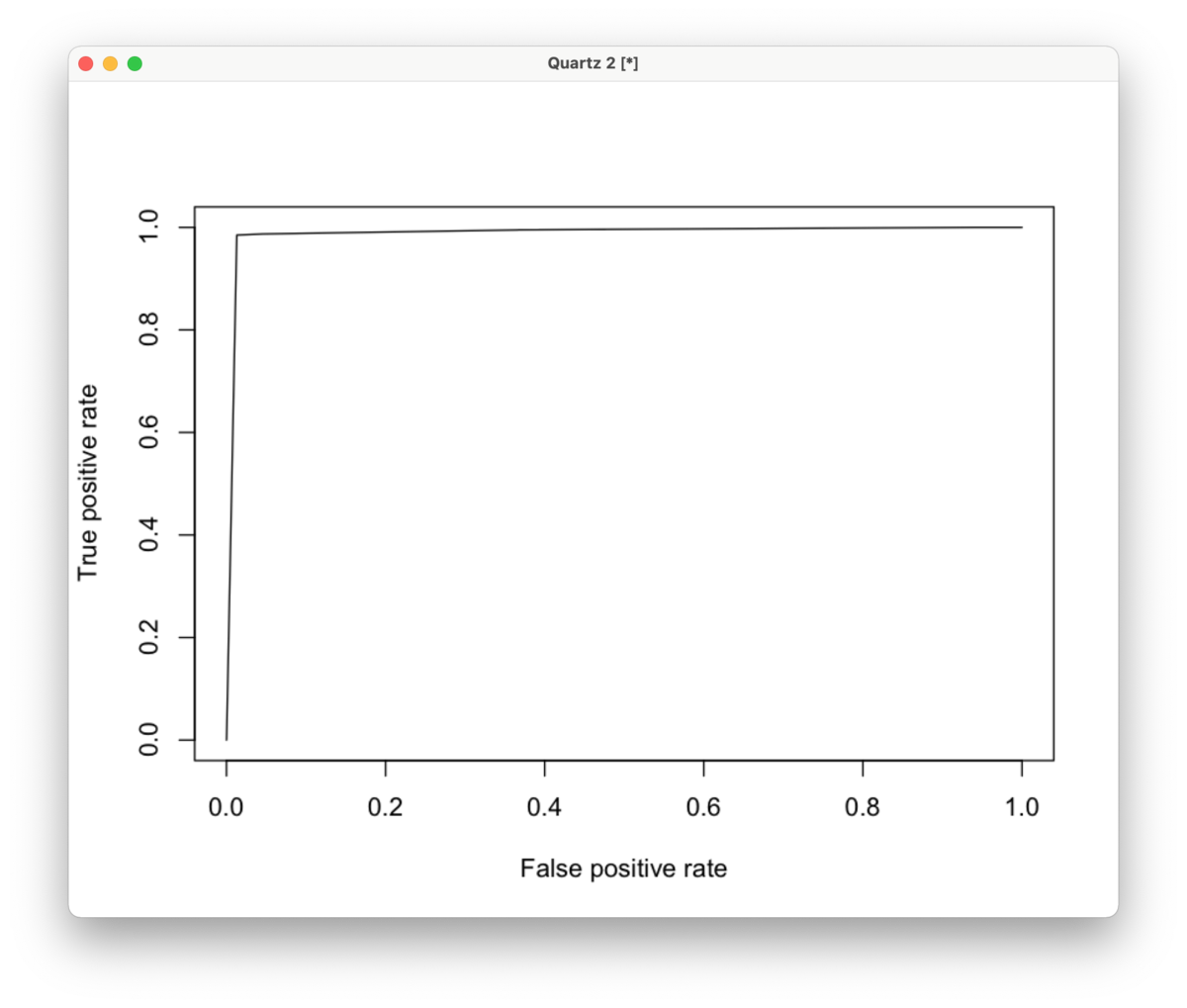
AUC:0.9886305
※下記Rコード
data1 <- cbind(x=rnorm(100000)*100, y=rnorm(100000)*100, flag=0)
data2 <- cbind(x=rnorm(1000)*5+30, y=rnorm(1000)*5+30, flag=1)
data <- rbind(data1, data2)
#plot(data[, 1], data[, 2], col=data[, 3]+1, pch=20, xlim=c(-100, 100), ylim=c(-100, 100))
library(rpart)
library(rpart.plot)
library(partykit)
library(ROCR)
tree <- rpart(factor(data[, 3]) ~ data[, 1] + data[, 2])
plot(tree)
text(tree, use.n = TRUE)
rpart.plot(tree)
plot(as.party(tree), gp=gpar(fontsize=9))
pred <- predict(tree)[(nrow(data)+1):(nrow(data)*2)]
pred1 <- prediction(pred, data[, 3])
perf <- performance(pred1,"tpr","fpr")
plot(perf)
auc.tmp <- performance(pred1,"auc")
auc <- as.numeric(auc.tmp@y.values)
auc
data.sample <- rbind(data1[sample(1:100000, 1000), ], data2)
#plot(data.sample[, 1], data.sample[, 2], col=data.sample[, 3]+1, pch=20, xlim=c(-100, 100), ylim=c(-100, 100))
tree.sample <- rpart(factor(data.sample[, 3]) ~ data.sample[, 1] + data.sample[, 2])
plot(as.party(tree.sample), gp=gpar(fontsize=9))
pred <- predict(tree.sample)[(nrow(data.sample)+1):(nrow(data.sample)*2)]
pred1 <- prediction(pred, data.sample[, 3])
perf <- performance(pred1,"tpr","fpr")
plot(perf)
auc.tmp <- performance(pred1,"auc")
auc <- as.numeric(auc.tmp@y.values)
auc
データサイエンティストからCDOへ
今年でiAnalysis社(アイアナ)を立ち上げてからちょうど10年経ちました。分析事業を大きくしたいと思って活動していたものの、資金調達などはできずに、結局は自分個人のコンサルティングに終始した10年でした。そういう意味ではうまくいかなかったなと思うものの、ちょうど今年から、クレディセゾン社のCDO(Chief Data Officer、最高データ責任者)に就くことになりました。セゾンへは去年からデータ分析のアドバイザーをしています。
セゾンのアドバイザーをする前は、ドコモ社に何年かコンサルティングをしていました。ドコモのデータ分析部署の立ち上げ時に協力し、顧客のLTV(Life Time Value)の開発をしたり、顧客体験の分析などをしていました。このときのドコモの部長に誘って頂いた勉強会でセゾンの役員と知り合い、アドバイザーをすることになりました。セゾンではいくつかのプロジェクトを見て、データの活用方法を改善することで、すぐに成果を出すことができ、CDOをオファーされた形です。いまもセゾン社内のいろんなチームを見ています。
自分はもともとデータサイエンティストと呼ばれることに抵抗感がありました。データサイエンティストと言うと、すべてについてデータを根拠にしないと許せないようなイメージを感じ、自分は違うのになと。むしろ、よく対比される勘と経験の方がしっくりくるタイプ。抽象的な絵も描くし麻雀やスロットなども好きで、アーティストな性格の方が合います。ドコモのプロジェクトが終わった後、何年か、絵を描きながら自分のやりたい事を考えていました。
アイアナを作ったのは、自分で事業をやりたいからで、それは今も変わってません。いつか大きい事業をやりたい。ただ、特に事業のネタがあるわけじゃない今、仕事として自分がやりたい事は何かなって考えると、経営者をやりたいなというところに行きつきました。それからは周りの人にアドバイスをもらいながら事業について考えつつ、セゾンで成果を出し、CDOになることができました。データサイエンティストはしっくりこないとは思いつつも、大学やコンサルで長年経験を積んできた専門分野ではあるので、それを活かして経営側に関わることが出来るのはとても有り難く感じています。個人的にはこれからの大企業を良くしていきたい、ひいては日本を良くしたいという思いもあります。CDOの役割を果たしつつ、経営者としての能力を磨き、日本の今後に少しでも役立てたらと思っています。